David Olivieri realizou un estudo sobre a inoculación só con porcións de virus
Conxugación de informática e inmunoloxía para afinar o éxito das vacinas nos animais
A base de datos resultante pode axudar a unha maior individualización dos tratamentos
Cando se vacina a un animal contra unha enfermidade estáselle inoculando, dun xeito atenuado, o propio virus, para que o seu sistema inmune “o memorice” e a próxima vez que se teña que enfrontar a el “en serio” poida defenderse axeitadamente. Non entanto, os programas de vacinación non están totalmente exentos de riscos para a saúde dos animais, o seu éxito non está garantido e teñen uns custes económicos significativos. Buscando melloras neste eido, David Olivieri, investigador do Grupo MILE da Universidade de Vigo, vén de realizar un estudo no que, conxugando informática e inmunoloxía, quere axudar a avanzar no eido das vacinas a medida para a gandería.
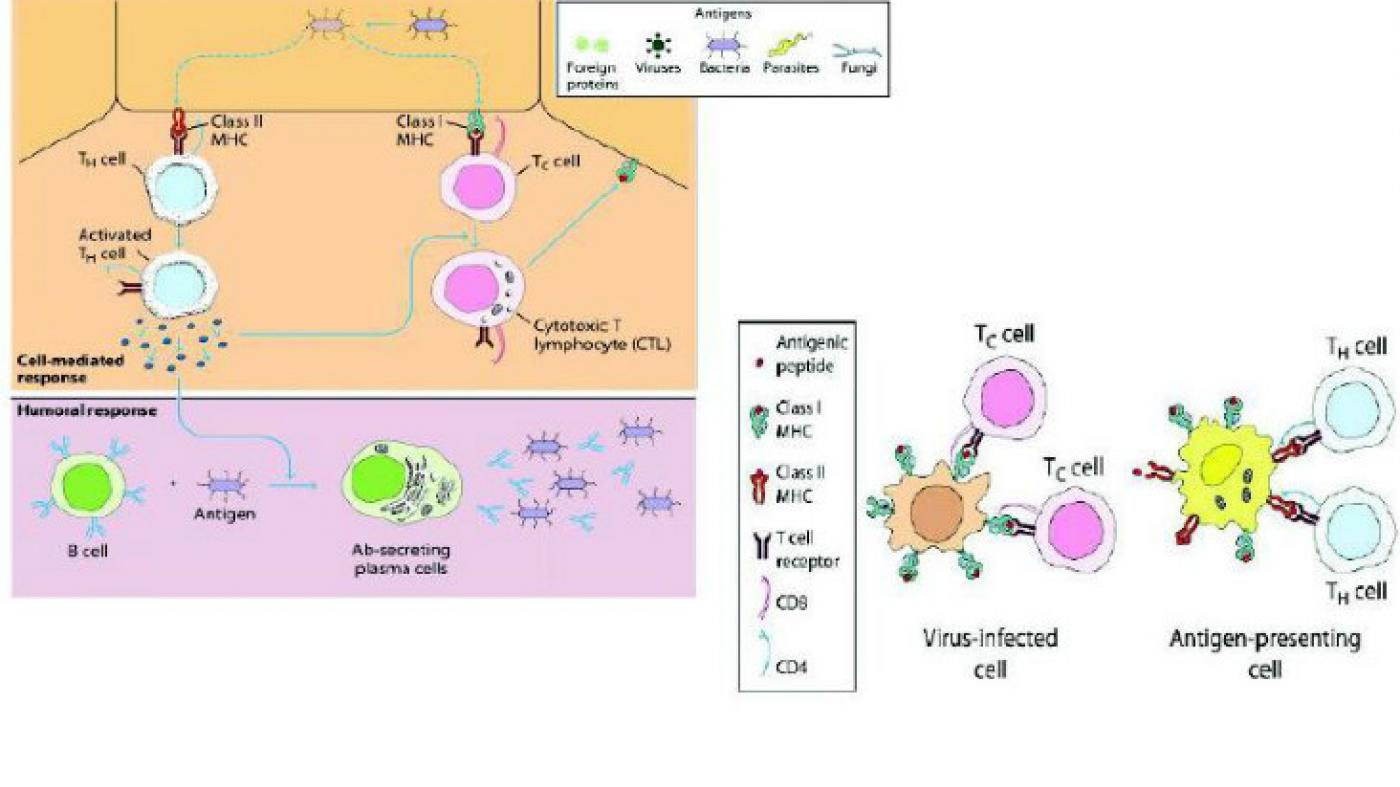
“A gran pregunta sobre a que traballamos é como podemos estimular o sistema inmune contra un virus sen introducir no animal, aínda que atenuado, ese patóxeno que produce a enfermidade?. Unha das posibles respostas é vacinando só cun anaco dese virus, cunha porción que é recoñecida polo sistema inmune. Isto faise a través de vacinas baseadas en epítopos, que estimulan e activan os linfocitos T, que á súa vez axudan na resposta inmune. É dicir, os epítopos son unha porción do antíxeno dunha macromolécula que se une cunha molécula do sistema inmunolóxico que se chama MHC para formar un complexo macromolecular que pode activar as citadas células T”, explica este físico e docente da Escola Superior de Enxeñaría Informática do campus de Ourense. O deseño das vacinas "a medida", comenta Olivieri, está baseado en atopar os epítopos dun virus que teñen unha alta afinidade para o conxunto de MHC presentes nunha raza ou poboación específica. As vacinas baseadas nestes pequenos epítopos, en lugar de utilizar o virus enteiro permiten que os linfocitos T do sistema inmune poidan ser activados igualmente para combater o virus e permitir así reducir o risco engadido para o animal.
Diferentes razas e diferentes patóxenos
O obxectivo do proxecto, que contou coa participación do biólogo da Universidade de Vigo José Faro e do investigador Iván Gómez, foi desenvolver unha ferramenta inmuno-informática e unha infraestrutura de base de datos de posible aplicación para razas especificas da gandería xa que permite, grazas á aplicación de diferentes algoritmos de intelixencia artificial, predicir epítopos para variantes de MHC existentes nesas razas. Ante a imposibilidade de ter datos xenómicos da gandería ourensá e galega, para a elaboración desta base de datos escolléronse cepas que puideran ser semellantes ás existentes en Ourense.Como “proba do concepto”, utilizouse o virus da lingua azul, que afecta a rumiantes como vacas e ovellas, para identificar epítopos para unhas razas de gandería similares ás da provincia de Ourense. “Por un lado, os virus mutan transformándose en diferentes cepas e, por outro, en diferentes zonas as poboacións de animais teñen diferenzas xenéticas (e polo tanto diferentes MHC). Isto provoca que unha vacina non vai a ser igualmente efectiva para todos. Unha vacina tradicional intenta dar unha única resposta dando unha mellor cobertura a todas as razas no mesmo momento. O obxectivo da tecnoloxía é avanzar no deseño dunha vacina que a pesar desta variabilidade ofreza unha solución personalizada para unha cepa ou poboación”, comenta o responsable do estudo, que contou cunha axuda INOU da Vicerreitoría do campus e da Deputación Provincial.
Destacando o apaixonante que é a investigación do sistema inmunolóxico, David Olivieri salienta que a idea chave das vacinas personalizadas é a selección racional de epítopos individuais. Deste xeito, un obxectivo principal do campo da inmunómica computacional, aplicada neste proxecto, é a identificación dos péptidos que se poden ligar ás moléculas MHC, que están presentes na superficie de calquera célula con núcleo, non só nas linfáticas, e que presentan antíxeno aos linfocitos T, permitindo activar o sistema inmunolóxico empregando non un virus completo senón só unha parte del. O resultado do estudo realizado, indica o seu responsable, podería ser empregado para motivar futuros estudos máis detallados e para establecer un mapa de polimorfismo MHC da gandería na provincia de Ourense, co fin de optimizar as vacinas para animais locais.